技術介紹:宏基因組測序技術與結果展示
宏基因組(Metagenomics),也稱元基因組,利用新一代高通量測序技術( NGS)以特定環境下微生物群體基因組為研究對象,在分析微生物多樣性、種群結構、進化關系的基礎上,可進一步探究微生物群體功能活性、相互協作關系及與環境之間的關系,發掘潛在的生物學意義。與傳統微生物研究方法相比,宏基因組測序技術規避了絕大部分微生物不能培養、痕量菌無法檢測的缺點,因此近年來在環境微生物學研究中得到了廣泛應用。
技術優勢
通量高,擁有標準化實驗室和高通量測序平臺,數據庫可靠
可檢測不可培養物種,可檢測痕量微生物
專業的生物信息團隊,可以滿足個性化的生物信息分析要求
技術路線
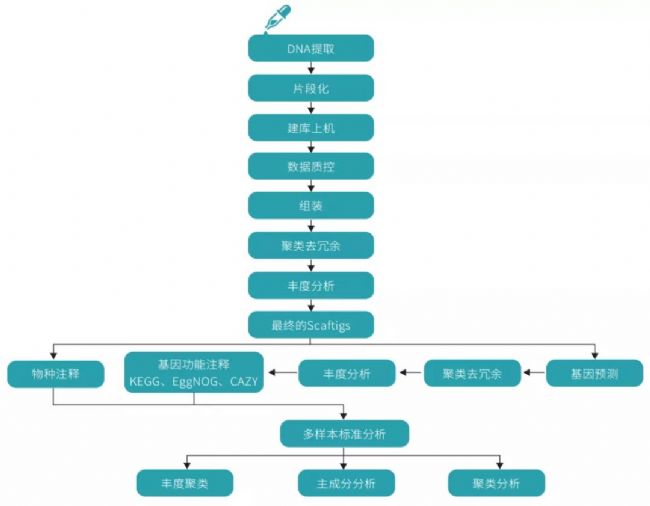
技術參數
樣本要求
土壤:10 g/sample
糞便:3-5 g/sample
血液:10 mL/sample
污泥/沉積物:5-10 g/sample
DNA:總量≥1 ug,濃度≥100 ng/uL
檢測平臺
測序平臺:Illumina Nova Seq 6000
測序方法:PE150
測序深度:6G/10G/12G
常規項目周期
實驗檢測::21個自然日
數據分析:21個自然日
應用方向
1.醫學領域:代謝病研究、腫瘤癌癥研究等
2.畜牧領域:腸道、瘤胃(如產甲烷菌類群)與動物健康/營養消化研究等
3.農業領域:根據微生物與植物互作、農業耕作/施肥處理與土壤微生物群落研究等
4.環境領域:霧霾處理、污水治理、石油降解、酸性礦水處理及海洋環境研究等
5.生物能源:特殊功能的菌株、基因挖掘、工程菌的開發研究
6.特殊極端環境:極端環境條件下的微生物類群研究
案例分析

宏基因組和代謝組分析揭示腸道菌群明顯的結直腸癌階段性特異表型
研究對象:人
期刊:Nature Medicine
影響因子:36.13
時間:2019年
1.研究背景
大多數散發性結直腸癌的發生是多步過程,首先形成息肉樣腺瘤,然后發展為粘膜內癌,最后發展為惡性腫瘤,其形成需要歷經幾十年的時間,早期癌癥的發現和內鏡切除是癌癥控制有效手段,腸道菌群與結直腸癌的發生有密切關系。
2.研究結果
基于宏基因組和代謝組研究平臺,對來自大隊列CRC中的616名和406名樣本分別進行了糞便宏基因組和代謝組學研究,獲得不同階段(MP,S0,Sl /SIl,II/SIV,HS)病例特異性表型的微生物(Atopobiumparvulum和B.wadsworthia等)和代謝(氨基酸和膽汁酸等)標志物;且進一步探索個體直腸癌患者的腸道微生物組與腫瘤與特征之間的關系。
3.結果展示
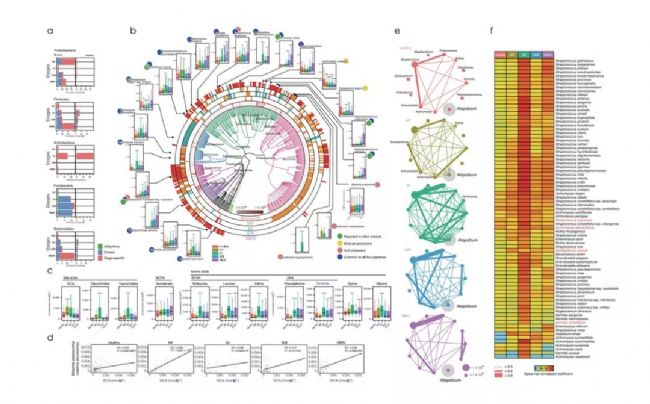
(1)宏基因組和代謝組分析
宏基因組分析首先發現Bacteroides和Prevotella是所有受試者和健康對照中變化最大的菌。進一步的研究還確定了與CRC關聯的新物種,其中Colinsella aerofaciens,Dorealongicatena,Porphyro-monasuenonis和Streptococcus anginosus等在III/IV中顯著升高。代謝組分析發現,大腸中主要能源物質丙酸鹽和丁酸鹽是含量最高的兩種代謝物,二氫尿嘧啶和尿素也存在較大差異。與健康對照組相比,MP中DCA(脫氧膽酸鹽)濃度顯著升高;SO中甘氨膽酸鹽和牛磺膽酸鹽濃度顯著升高;支鏈氨基酸,苯丙氨酸,酪氨酸,甘氨酸,絲氨酸濃度也升高。
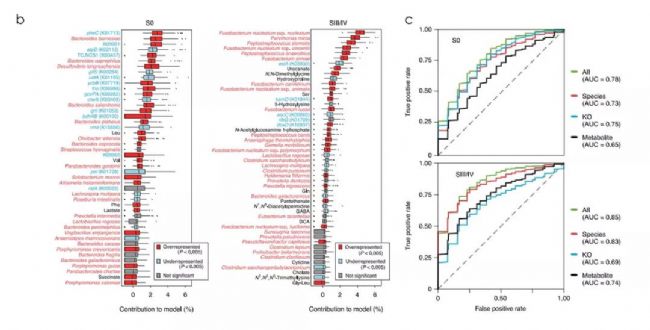
(2)功能分析和標志物篩選
基于KEGG數據庫篩選出1243個同源基因(KO基因)在至少一個階段顯著升高,96個KO基因在至少一個階段顯著降低。在差異最顯著的通路中,芳香族氨基酸代謝和產硫化物的通路與CRC相關。參與苯丙氨酸和酪氨酸合成的基因顯著升高,其中pheC被確定為區分S0患者和健康對照的得分最高標志物。通過構建隨機森林和LASSO邏輯回歸模型來區分健康對照與SO和SIII/IV。在S0分類中,特征值是包括編碼環己二烯基脫水酶的pheC的KO基因;在SIII/IV分類中,特征值為已被確認為CRC標志物的P.micraP.stomatis和 F.nucleatum等口腔厭氧菌。
(3)結果驗證和潛在機制
對28名CRC患者(SI /II和SIII /IV)術前和術后1年的糞便樣品進行宏基因組測序。結果表明,腫瘤切除后P.stomatis,P.anaerobius和P.uenonis的相對豐度降低。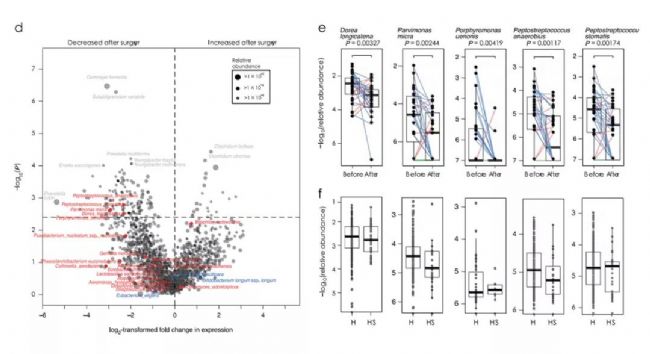
參考文獻
Yachida S et al., Metagenomic and metabolomic analyses reveal distinct stage-specific phenotypes of the gut microbiota in colorectal cancer.Nat Med.2019 doi: 10.1038/s41591-019-0458-7.
技術優勢
通量高,擁有標準化實驗室和高通量測序平臺,數據庫可靠
可檢測不可培養物種,可檢測痕量微生物
專業的生物信息團隊,可以滿足個性化的生物信息分析要求
技術路線

技術參數
樣本要求
土壤:10 g/sample
糞便:3-5 g/sample
血液:10 mL/sample
污泥/沉積物:5-10 g/sample
DNA:總量≥1 ug,濃度≥100 ng/uL
檢測平臺
測序平臺:Illumina Nova Seq 6000
測序方法:PE150
測序深度:6G/10G/12G
常規項目周期
實驗檢測::21個自然日
數據分析:21個自然日
應用方向
1.醫學領域:代謝病研究、腫瘤癌癥研究等
2.畜牧領域:腸道、瘤胃(如產甲烷菌類群)與動物健康/營養消化研究等
3.農業領域:根據微生物與植物互作、農業耕作/施肥處理與土壤微生物群落研究等
4.環境領域:霧霾處理、污水治理、石油降解、酸性礦水處理及海洋環境研究等
5.生物能源:特殊功能的菌株、基因挖掘、工程菌的開發研究
6.特殊極端環境:極端環境條件下的微生物類群研究
案例分析

宏基因組和代謝組分析揭示腸道菌群明顯的結直腸癌階段性特異表型
研究對象:人
期刊:Nature Medicine
影響因子:36.13
時間:2019年
1.研究背景
大多數散發性結直腸癌的發生是多步過程,首先形成息肉樣腺瘤,然后發展為粘膜內癌,最后發展為惡性腫瘤,其形成需要歷經幾十年的時間,早期癌癥的發現和內鏡切除是癌癥控制有效手段,腸道菌群與結直腸癌的發生有密切關系。
2.研究結果
基于宏基因組和代謝組研究平臺,對來自大隊列CRC中的616名和406名樣本分別進行了糞便宏基因組和代謝組學研究,獲得不同階段(MP,S0,Sl /SIl,II/SIV,HS)病例特異性表型的微生物(Atopobiumparvulum和B.wadsworthia等)和代謝(氨基酸和膽汁酸等)標志物;且進一步探索個體直腸癌患者的腸道微生物組與腫瘤與特征之間的關系。
3.結果展示
(1)宏基因組和代謝組分析
宏基因組分析首先發現Bacteroides和Prevotella是所有受試者和健康對照中變化最大的菌。進一步的研究還確定了與CRC關聯的新物種,其中Colinsella aerofaciens,Dorealongicatena,Porphyro-monasuenonis和Streptococcus anginosus等在III/IV中顯著升高。代謝組分析發現,大腸中主要能源物質丙酸鹽和丁酸鹽是含量最高的兩種代謝物,二氫尿嘧啶和尿素也存在較大差異。與健康對照組相比,MP中DCA(脫氧膽酸鹽)濃度顯著升高;SO中甘氨膽酸鹽和牛磺膽酸鹽濃度顯著升高;支鏈氨基酸,苯丙氨酸,酪氨酸,甘氨酸,絲氨酸濃度也升高。

圖1.腸道菌群與代謝物變化
(2)功能分析和標志物篩選
基于KEGG數據庫篩選出1243個同源基因(KO基因)在至少一個階段顯著升高,96個KO基因在至少一個階段顯著降低。在差異最顯著的通路中,芳香族氨基酸代謝和產硫化物的通路與CRC相關。參與苯丙氨酸和酪氨酸合成的基因顯著升高,其中pheC被確定為區分S0患者和健康對照的得分最高標志物。通過構建隨機森林和LASSO邏輯回歸模型來區分健康對照與SO和SIII/IV。在S0分類中,特征值是包括編碼環己二烯基脫水酶的pheC的KO基因;在SIII/IV分類中,特征值為已被確認為CRC標志物的P.micraP.stomatis和 F.nucleatum等口腔厭氧菌。

圖2.標志物篩選
(3)結果驗證和潛在機制
對28名CRC患者(SI /II和SIII /IV)術前和術后1年的糞便樣品進行宏基因組測序。結果表明,腫瘤切除后P.stomatis,P.anaerobius和P.uenonis的相對豐度降低。

圖3.與CRC相關微生物和代謝物驗證結果
參考文獻
Yachida S et al., Metagenomic and metabolomic analyses reveal distinct stage-specific phenotypes of the gut microbiota in colorectal cancer.Nat Med.2019 doi: 10.1038/s41591-019-0458-7.
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com